ANCIENT DNA - GENE AUS ALTER ZEIT.
Über die molekularbiologische Analyse archäologischer Funde
Vor mittlerweile 14 Jahren (Audic et Beraud-Colomb, 1997) wurde die Archäometrie durch eine neue Disziplin bereichert. Molekularbiologen konnten "alte DNA" in ägyptischen Mumien (Pääbo, 1985) und in den Knochen eines ausgestorbenen Pferdeverwandten (Quagga) (Higuchi et al., 1984) nachweisen.
Damit war eine weitverbreitete Lehrmeinung widerlegt, die davon ausging, daß DNA (Desoxyribonukleinsäure, Träger der Erbinformation) schon kurz nach dem Tod eines Organismus so weit abgebaut sei, daß keinerlei intakte genetische Information mehr vorhanden wäre. Es hat sich aber gezeigt, daß alte DNA (aDNA) zwar stark degradiert, aber in vielen Fällen immer noch genug Erbinformation für eine Analyse erhalten ist.
In den Frühzeiten der Molekularen Archäologie war man auf vergleichsweise große Mengen an DNA angewiesen, da die damals gebräuchlichen Analysemethoden nicht sensitiv genug waren um Spuren nachzuweisen. Mitte der achtziger Jahre wurde die Polymerase-Kettenreaktion (PCR, Polymerase-Chain-Reaction) entwickelt. Dies stellte für die gesamte Molekularbiologie und Gentechnik einen Quantensprung in der Methodik dar: mit der PCR ist es möglich, geringste Mengen von Erbsubstanz nachzuweisen. Die Polymerase-Kettenreaktion basiert auf einer hochspezifischen Anreicherung einer Ziel-DNA, die dann leicht mit konventionellen Methoden der Molekularbiologie ausgewertet werden kann; theoretisch reicht eine einzige intakte Einheit eines DNA-Abschnittes für einen positiven Nachweis (Krawczak, 1994).
Archäologische Fragen und genetische Information:
Eine archäologische Fragestellung kann dann molekularbiologisch beantwortet werden, wenn sie sich auf eine Eigenschaft bezieht, die in der Erbinformation festgelegt ist:
Im einfachsten Fall ist dies eine Geschlechtsbestimmung (Herrmann et Hummel, 1994): Hier wird der Umstand ausgenützt, daß für manche Gene eine weibliche und eine männliche Variante existiert, die auf dem X- bzw. dem Y-Chromosom (Geschlechts-chromosomen) lokalisiert ist. Kann nur die weibliche Variante nachgewiesen werden, so handelt es sich um ein weibliches Individuum (zwei X-Chromosomen); können hingegen beide Varianten nachgewiesen werden, so ist das Individuum männlich (ein X- und ein Y-Chromosom). Auf diese Weise kann ein anthropologischer Befund bestätigt und ergänzt werden. Weiters ermöglicht eine DNA-Analyse auch dann eine Geschlechtsbestimmung, wenn aufgrund starker Zerstörungen oder unvollständiger Funde keine morphometrische Bestimmung mehr möglich ist.
Darüber hinaus kann eine DNA-Analyse auch zur Bestimmung der Verwandtschaftsverhältnisse von Individuen herangezogen werden. Die aus der forensischen Medizin entlehnte Methode des "DNA-Fingerprinting" ermöglicht die Erstellung eines DNA-Profils, das wie konventionelle Fingerabdrücke eine Zuordnung erlaubt. Weiters gehorchen bestimmte DNA-Profile den Gesetzen der Mendelschen Vererbungslehre, was die Rekonstruktion einer Genealogie (z.B. in einem Gräberfeld oder einer Gruft; etc.) ermöglicht. Weiters ist es mit DNA-Fingerprints auch möglich, einzelne, verstreut liegende Skelettelemente zuzuordnen (Schultes et al., 1997) oder auch Leichenfunde zu identifizieren (Mengele (Jeffreys et al., 1992), Romanov (Gill et al. 1994)).
Wiederum andere Genstrukturen erlauben die Bestimmung eines übergeordneten Populationskontextes für ein Individuum, wie es u.a. auch für die Leiche des Mannes vom Hauslabjoch (Handt et al. 1994) durchgeführt wurde. Diese vergleichende Studie ergab die höchste Übereinstimmung mit Gensequenzen der heutigen Bevölkerung der Nordalpen. Unter diesem populationsgenetischen Gesichtspunkt wurden auch zahlreiche andere Fundstellen bzw. Gräberfelder untersucht, hinsichtlich der Erforschung von Entwicklung, Wanderung und Verteilung verschiedener alter, aber auch rezenter Populationen und Völker. An dieser Stelle sollen auch jene Arbeiten erwähnt sein, die sich mit Modellen der Homo-Evolution auseinandersetzen, welche auf genetischen Daten basieren.
Neben den "klassischen" biologischen Methoden zur Bestimmung einer Spezies werden in zunehmendem Maße auch genetische Daten zur Systematisierung herangezogen. Vergleichende DNA-Sequenz-Analysen von tierischem und pflanzlichem Material, welches mit einem Fund assoziiert ist, ermöglichen die Rekonstruktion der Ernährungsgewohnheiten einer alten Zivilisation oder auch einen Einblick in die Artenvielfalt vergangener Zeiten (Rollo et al., 1994; Allaby et al., 1997).
Das selbe Prinzip wurde für die Identifikation von Tuberkelbakterien (Erreger der Lungentuberkulose) in 500 bis 700 Jahre alten peruanischen (Arriaza et al., 1995; Morell, 1994) und 2500 Jahre alten ägyptischen (Nehrlich et al., 1997) Mumien verwendet. Somit sollte es möglich sein, jede genetische Manifestation einer Krankheit nachzuweisen. Das trifft sowohl für Fremdorganismen (mikrobielle Pathogene) als auch für Erbkrankheiten zu.
Quellenmaterial
Die Wiederauferstehung der Dinosaurier wird wohl Utopie bleiben - Realität hingegen sind genetische Daten von Insekten und Pflanzen, die vor Jahrmillionen in Bernstein eingeschlossen wurden (Herrmann et Hummel, 1994). Allerdings stellt Bernstein nur eine von vielen DNA-Quellen dar. Abgesehen von solchen Glücksfällen wie vollständig erhaltene Mumien sind vor allem Zähne und Knochen für DNA-Analysen besonders geeignet, da diese harten Gewebe gute Voraussetzungen für den Erhalt intakter genetischer Information bieten. Knochen sind von vielen Hohlräumen durchsetzt, die im lebenden Organismus Zellen enthalten, die den Knochen auf-, ab- und umbauen. Beim Tod des Individuums durchlaufen diese Zellen eine natürliche individuelle Mumifikation.
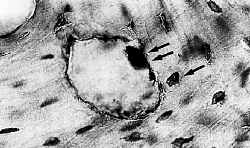
Abb. 1:
Histologischer Schnitt eines mittelalterlichen
Knochens, Pfeile markieren Zellüberreste mit gefärbter DNA (aus: Herrmann, B. and S. Hummel, Eds. (1994). Ancient DNA. Recovery and Analysis of Genetic Material from Palaeontological, Archaeological, Museum, Medical and Forensic Specimens, 8)
|
Nebenstehende Abbildung (Abb. 1) zeigt einen mikroskopischen Dünnschnitt eines mittelalterlichen Knochens, der mit einer DNA-spezifischen Färbemethode behandelt wurde. Demnach sind die dunklen Flecken Überreste von Knochenzellen, deren DNA für die Färbung verantwortlich ist.
Prinzipiell sind alle biogenen Überreste für eine molekular-biologische Untersuchung geeignet. Das Spektrum an Quellenmaterialien reicht von Knochen, Zähnen und Weichgeweben über Samen, Mikroorganismen und Fossilien hin bis zu medizinischen, forensischen und musealen Präparaten, um nur einige zu nennen. Sogar in verkohlten Weizensamen (Allaby et al., 1997) und in den Farben einer Höhlenmalerei (Service, 1995) konnte noch intakte genetische Information gefunden werden.
|
Obwohl sich schon viele Studien mit diesem Thema beschäftigt haben, konnte noch keine befriedigende Methode gefunden werden, mit der im Vorfeld festgestellt werden kann, ob das Fundstück eine ausführlichere molekularbiologische Untersuchung wert ist. Für den Erhaltungszustand der DNA ist das Alter sicher kein Maß. Sondern das Liegemilieu und diagenetische Umwelteinflüsse (Burger et al., 1997): Trockenheit und Hitze, wie sie in Wüstenklima-Regionen zu finden sind oder auch große Kälte (Gletscher, Polarregionen, etc.) sind hervorragende Kriterien für die Erhaltung genetischer Information. Generell begünstigen den Erhalt von DNA neben einem neutralen pH-Wert des Liegemilieus vor allem Bedingungen, unter denen kein oder überhaupt kein Stoffwechsel möglich ist, wobei z.B. auch erdgelagerte Knochen und Zähne mit hoher Wahrscheinlichkeit auch dann noch DNA enthalten, wenn sie schon mehrere tausend Jahre alt sind.
Praktische Aspekte bei der Probenahme (Herrmann et Hummel, 1994, Kapitel 4): Wie bereits angedeutet können alle möglichen Arten von biogenen Überresten für eine Gen-Analyse geeignet sein. Selten aber ist es möglich, eine Probe unter optimalen Bedingungen zu nehmen.
Was für andere analytische Verfahren gilt, das ist insbesondere für die molekularbiologische Analytik von Bedeutung: die Vermeidung von Kontaminationen mit Fremd-DNA. Eine Berührung mit der bloßen Hand reicht aus um ein Objekt zu kontaminieren, geschweige Husten oder Niesen.
Um das Risiko einer solchen Verunreinigung zu minimieren, sollte die Probenahme mit Untersuchungshandschuhen, eventuell einem Mundschutz und sterilem Werkzeug wenn möglich noch am Fund- bzw. Ausgrabungsort stattfinden. Die Proben sind sofort in luftdichten Behältnissen zu verstauen und bis zur Öffnung im Labor so zu lagern, daß keine weiteren Kontaminationen mehr auftreten und die DNA nicht weiter geschädigt wird.
Meist aber sind die Proben unter suboptimalen Bedingungen genommen worden und durch viele Hände gegangen, sodaß vor dem Aufschluß die Probe gründlich gereinigt werden muß; wenn möglich sollte die Oberfläche abgetragen werden. Darüber hinaus sind eine Reihe von Kontrollexperimenten durchzuführen, um die Authentizität alter genetischer Information zu gewährleisten.
"Konventionelle" Methoden aus dem Bereich der chemisch-physikalischen Analytik (Radiocarbondatierung, Röntgenaufnahmen, Computertomographien, etc.) haben sich als ein fester Bestandteil der archäometrischen Datenerfassung etabliert. Wenn biogenes Material zur Verfügung steht, so ist die Molekularbiologie instande, einen wesentlichen Beitrag zu leisten: anthropologische und archäologische Befunde können abgesichert werden und manchmal kann eine DNA-Analyse auch völlig neue Horizonte eröffnen.
Danksagung an Sven Buchholzer für orthographische Unterstützung.
Literatur:
Audic, S. and E. Beraud-Colomb (1997). „Ancient DNA is thirteen years old." Nat Biotechnol 15(9): 855-8. Paabo, S. (1985). „Molecular cloning of Ancient Egyptian mummy DNA." Nature 314(6012): 644-5. Higuchi, R., B. Bowman, et al. (1984). „DNA sequences from the quagga, an extinct member of the horse family." Nature 312(5991): 282-4. Krawczak, M. (1994). DNA-Fingerprinting. Heidelberg, Berlin, Oxford, Spektrum Akademischer Verlag. Herrmann, B. and S. Hummel, Eds. (1994). Ancient DNA. Recovery and Analysis of Genetic Material from Paläontological, Archaeological, Museum, Medical and Forensic Specimens. New York, Berlin, Heidelberg, Springer Verlag. Schultes, T., S. Hummel, et al. (1997). „[Classification of isolated skeletal elements using aDNA typing], in German." Anthropol Anz 55(2): 207-16. Jeffreys, A.J., M. J. Allen, et al. (1992). „Identification of the skeletal remains of Josef Mengele by DNA analysis." Forensic Sci Int 56(1): 65-76. Gill, P., P.L. Ivanov, et al. (1994). „Identification of the remains of the Romanov family by DNA analysis [see comments]." Nat Genet 6(2): 130-5. Handt, O., M. Richards, et al. (1994). „Molecular genetic analyses of the Tyrolean Ice Man." Science 264(5166): 1775-8. Rollo, F., W. Asci, et al. (1994). „Molecular Ecology of a Neolithic Meadow: the DNA of the grass remains from the archaeological site of the Tyrolean Iceman." Experientia 50: 576-584. Allaby, R.G., K. O´Donnoghue, et al. (1997). „Evidence for the Survival of Ancient DNA in Charred Wheat Seeds from European Archaeological Sites." Ancient Biomolecules 1 (2): 119-129. Arriaza, B.T., W. Salo, et al. (1995). „Pre-Columbian tuberculosis in northern Chile: molecular and skeletal evidence." Am J Phys Anthropol 98(1): 37-45. Morell, V. (1994). „Mummy settles TB antiquity debate [news]." Science 263(5154): 1686-7. Nerlich, A. G., C.J. Haas, et al. (1997). „Molecular evidence for tuberculosis in an ancient Egyptian mummy [letter; comment]." Lancet 350(9088): 1404. Service, R.F. (1995). „Rock Paintings Yield DNA." Science 268: 501. Burger, J., S. Hummel, et al. (1997). „[Detection of DNA single-copy sequences of prehistoric teeth. Site milieu as a factor for preservation of DNA]." Anthropol Anz 55(2): 193-8.
Weiterführende Literatur:
Herrmann, B., Ed. (1994). Archäometrie. Naturwissenschaftliche Analyse von Sachüberresten. Berlin Heidelberg, Springer Verlag. Herrmann, B. and S. Hummel, Eds. (1994). Ancient DNA. Recovery and Analysis of Genetic Material from Paläontological, Archaeological, Museum, Medical and Forensic Specimens. New York, Berlin, Heidelberg, Springer Verlag. Krawczak, M. (1994). DNA-Fingerprinting. Heidelberg, Berlin, Oxford, Spektrum Akademischer Verlag. Newton, C. R. and A. Graham (1994). PCR. Heidelberg, Berlin, Oxford, Spektrum Akademischer Verlag. Ancient Biomolecules, Journal, Harwood Academic publishers. Stryer, Lubert (1994). Biochemie. Spektrum Akademischer Verlag.
http://www.comic.sbg.ac.at/staff/jan/ancient/titel.htm
© Jan Kiesslich
e-mail: jan@comic.sbg.ac.at

